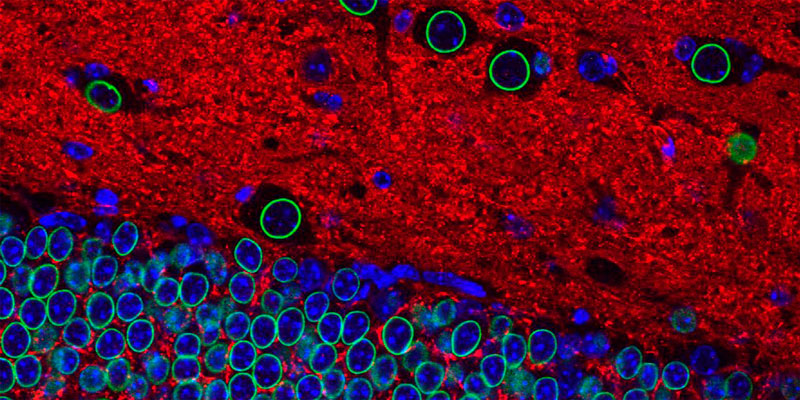
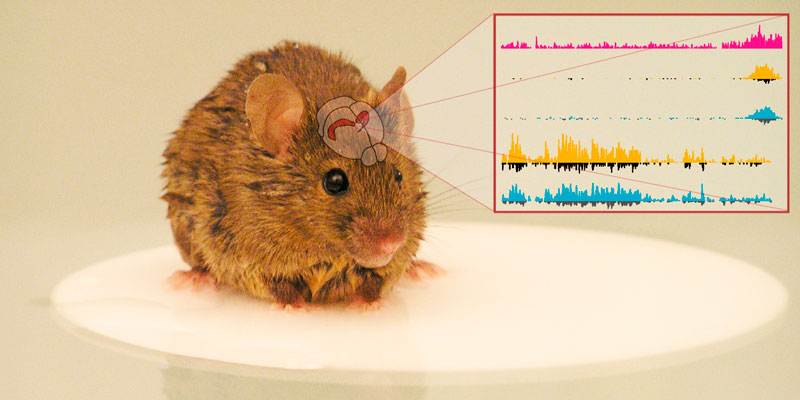
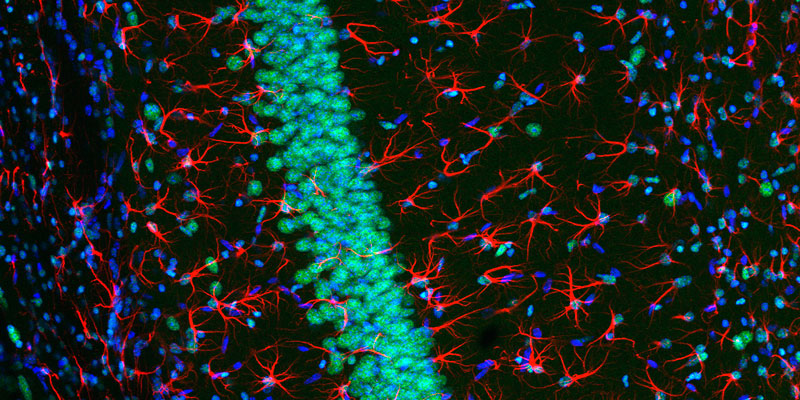
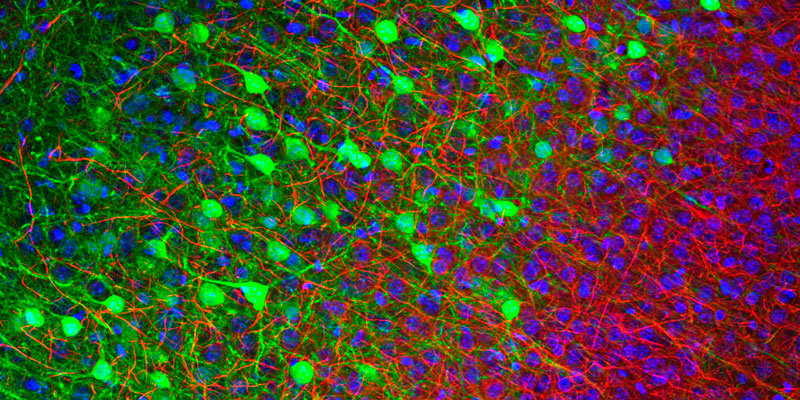
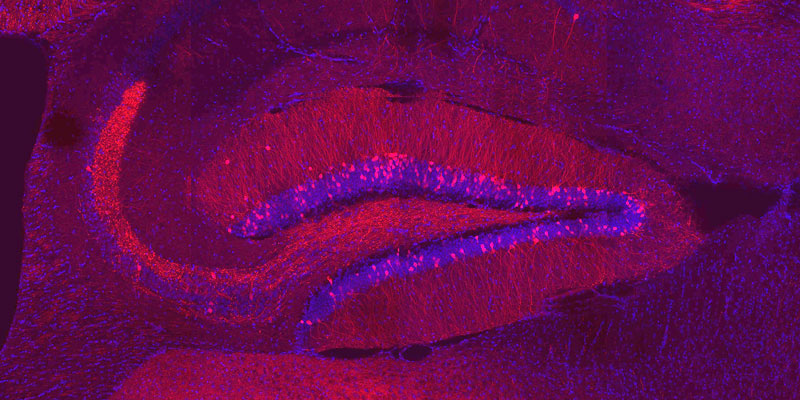
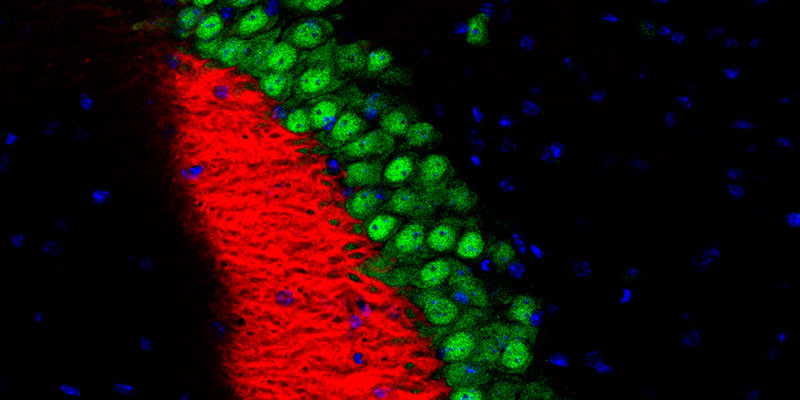
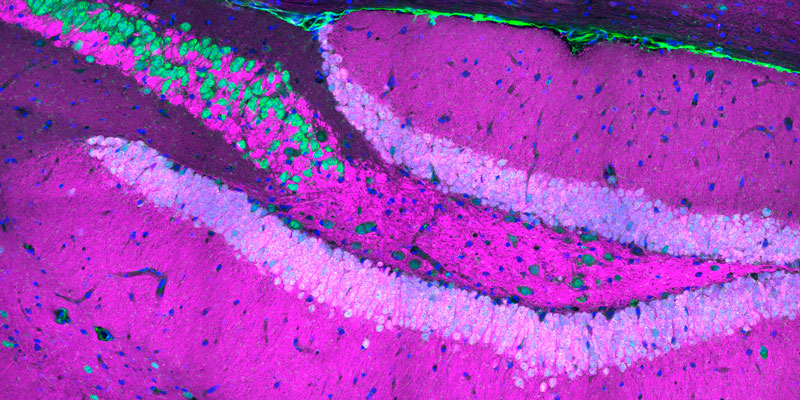
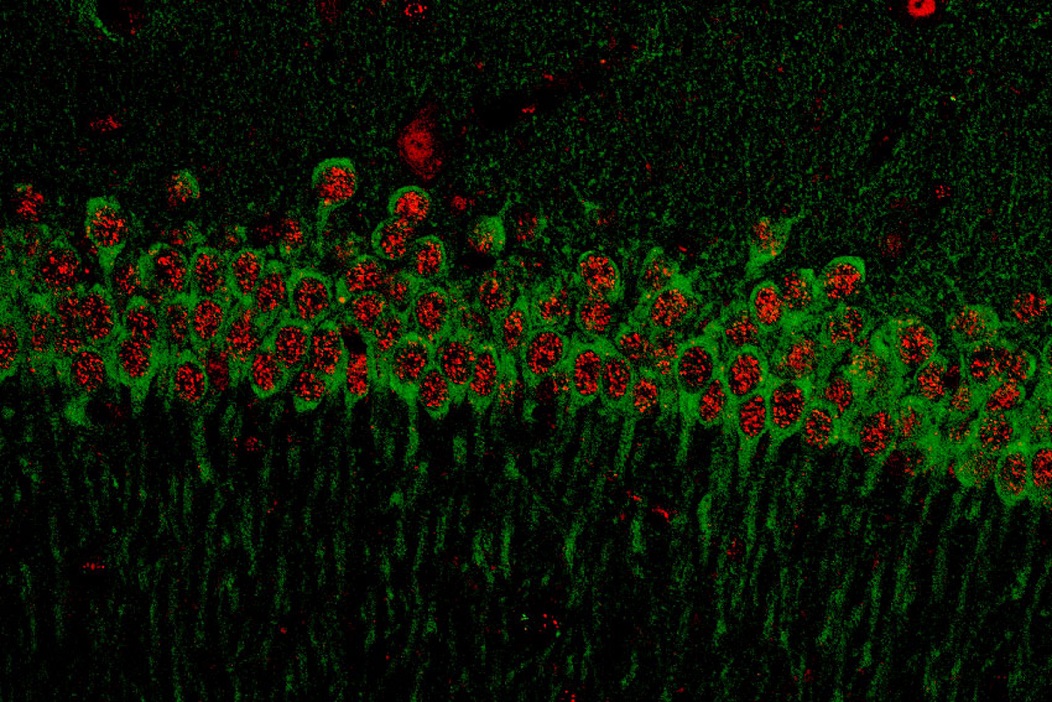
Líneas de investigación
Nuestra investigación se centra en los mecanismos moleculares que permiten el aprendizaje, la formación de nuevos recuerdos y otras modificaciones duraderas del comportamiento animal. En concreto, investigamos el papel de determinados factores de transcripción y epigenéticos en esos procesos. También investigamos cómo el mal funcionamiento de estos mecanismos puede dar lugar a patologías del sistema nervioso. Para abordar esas cuestiones usamos una aproximación multidisciplinar que combina estudios de genética, genómica, biología molecular y celular, electrofisiología y conducta animal. Desde el punto de vista metodológico, estamos particularmente interesados en la aplicación de las nuevas tecnologías de edición epigenética y perfilado genómico en el sistema nervioso.
Nuestra investigación actual se centra en las siguientes dos áreas:
• Regulación de la expresión génica dependiente de actividad por mecanismos epigenéticos y transcripcionales: Los modelos celulares actuales para explicar cómo se forman las memorias proponen que los recuerdos están codificados en forma de cambios en la fuerza de conexiones sinápticas específicas. Estos cambios requieren a su vez de cambios en la expresión génica de las neuronas. En el laboratorio, estamos interesados en explorar el papel que juegan en este proceso algunos factores de transcripción regulados por actividad (como CREB y SRF), algunas enzimas epigenéticas (como CBP y p300), y la modificación covalente de histonas y la metilación del DNA.
• Contribución de mecanismos epigenéticos a la patoetiología de la discapacidad intelectual: Investigamos la relación entre fallos en los mecanismos de regulación epigenética y diversos trastornos neurológicos asociados a problemas cognitivos que son hoy en día incurables, tales como el síndrome de Rubinstein-Taybi y la discapacidad intelectual asociada al cromosoma X. Para ello, generamos y caracterizamos modelos murinos de estos trastornos, investigamos las causas moleculares que subyacen a los síntomas y ensayamos nuevas terapias.
Publicaciones relevantes
- Kdm1a safeguards the topological boundaries of PRC2-repressed genes and prevents aging-related euchromatinization in neurons. Del Blanco B, Niñerola S, Martín-González AM, Paraíso-Luna J, Kim M, Muñoz-Viana R, Racovac C, Sánchez-Mut JV, Ruan Y and Barco A. Nature Communications. 2024 15, 1781 (2024) https://www.nature.com/articles/s41467-024-45773-3
- Targeting epigenetic dysregulation in autism spectrum disorders. Macarena L. Herrera, Juan Paraíso-Luna , Isabel Bustos-Martínez, Ángel Barco. Trends in Molecular Medicine. 2024 30(11): p1028-1046 https://doi.org/10.1016/j.molmed.2024.06.004
- CBP is required for establishing adaptive gene programs in the adult mouse brain. Lipinski, M.#, Niñerola, S#., Fuentes-Ramos, M., Valor, L.M., del Blanco, B., López-Atalaya, J.P., Barco, A. J Neurosci. 2022 42(42): 7984-8001 https://doi.org/10.1523/JNEUROSCI.0970-22.2022
- KAT3-dependent acetylation of cell type-specific genes maintains neuronal identity in the adult mouse brain Lipinski M, Muñoz-Viana R, Del Blanco B, Marquez-Galera A, Medrano-Relinque J, Caramés JM, Szczepankiewicz AA, Fernandez-Albert J, Navarrón CM, Olivares R, Wilczyński GM, Canals S, Lopez-Atalaya JP, Barco A Nat Commun 2020 11(1):2588 https://doi.org/10.1038/s41467-020-16246-0
- CBP/p300 in brain development and plasticity: disentangling the KAT’s cradle Lipinski M, Del Blanco B, Barco A Current Opinion in Neurobiology 2019 59:1 https://doi.org/10.1016/j.conb.2019.01.023
- Cbp-dependent histone acetylation mediates axon regeneration induced by environmental enrichment in rodent spinal cord injury models Hutson TH, Kathe C, Palmisano I, Bartholdi K, Hervera A, De Virgiliis F, McLachlan E, Zhou L, Kong G, Barraud Q, Danzi MC, Medrano-Fernandez A, Lopez-Atalaya JP, Boutillier AL, Sinha SH, Singh AK, Chaturbedy P, Moon LDF, Kundu TK, Bixby JL, Lemmon VP, Barco A, Courtine G, Di Giovanni S Sci Transl Med 2019 11(487):eaaw2064 https://doi.org/10.1126/scitranslmed.aaw2064
- Immediate and deferred epigenomic signatures of in vivo neuronal activation in mouse hippocampus Fernandez-Albert J, Lipinski M, Lopez-Cascales MT, Rowley MJ, Martin-Gonzalez AM, Del Blanco B, Corces VG, Barco A Nature Neuroscience 2019 22(10):1718 https://doi.org/10.1038/s41593-019-0476-2
- FrozONE: Quick Cell Nucleus Enrichment for Comprehensive Proteomic Analysis of Frozen Tissues. Huschet, LA, Kliem FP, Wienand P, Wunderlich C, Ribeiro A, Bustos-Martínez I, Barco A, Wunderlich FT, Lech M, Robles MS. Life Sci Alliance. 2025 8(3): e2024031301 https://doi.org/10.26508/lsa.202403130
- New Genome-wide Technologies to Study Activity-regulated Transcription. Niñerola S., Barco A. Chapter in “Regulation of transcription by neuronal activity: To the nucleus and back. 2nd Edition” 2024 (Springer Science Publishers). Pag: 561-591
- Unveiling Transcriptional and Epigenetic Mechanisms Within Engram Cells: Insights into Memory Formation and Stability. Fuentes-Ramos M., Barco A. Adv Neurobiol. 2024 38: 111-129 - Chapter in “Memory Engrams” (Springer Science Publishers) https://doi.org/10.1007/978-3-031-62983-9_7
- Targeting epigenetic dysregulation in autism spectrum disorders. Macarena L. Herrera, Juan Paraíso-Luna , Isabel Bustos-Martínez, Ángel Barco. Trends in Molecular Medicine. 2024 30(11): p1028-1046 https://doi.org/10.1016/j.molmed.2024.06.004
- Science around the world. Abel Plaza-Florido, Olga Gursky, Macarena Lorena Herrera, Charalampos D. Moschopoulos, Yahya Sohrabi Trends in Molecular Medicine. 2024 30(11): 997 - 1000 https://doi.org/10.1016/j.molmed.2024.09.007
- Keep calm and carry H3K27me1 off. Alcalá-Vida R, Barco A. Neuron 2024 112(17): 2829-2832 https://doi.org/10.1016/j.neuron.2024.07.014
- Kdm1a safeguards the topological boundaries of PRC2-repressed genes and prevents aging-related euchromatinization in neurons. Del Blanco B, Niñerola S, Martín-González AM, Paraíso-Luna J, Kim M, Muñoz-Viana R, Racovac C, Sánchez-Mut JV, Ruan Y and Barco A. Nature Communications. 2024 15, 1781 (2024) https://www.nature.com/articles/s41467-024-45773-3
- Extra centrosomes induce PIDD1‐mediated inflammation and immunosurveillance. Irmina Garcia‐Carpio, Vincent Z Braun, Elias S Weiler, Marina Leone , Sergio Niñerola, Angel Barco, Luca L Fava, Andreas Villunger. EMBO J. 2023 42: e113510 https://doi.org/10.15252/embj.2023113510
- Comprehensive expression analysis for the core cell cycle regulators in the chicken embryo reveals novel tissue-specific synexpression groups and similarities and differences with expression in mouse, frog and zebrafish Alaiz Noya M, Berti F, Dietrich S Journal of Anatomy. 2022 241 (1): 42-66 https://doi.org/10.1111/joa.13629
- CBP and p300 Jointly Maintain Neural Progenitor Viability but Play Unique Roles in the Differentiation of Neural Lineages. González-Martínez, R., Márquez-Galera, A., Del Blanco, B., López-Atalaya, J.P., Barco, A., Herrera, E. Cells 2022 11(24): 4118 https://doi.org/10.3390/cells11244118
- CBP is required for establishing adaptive gene programs in the adult mouse brain. Lipinski, M.#, Niñerola, S#., Fuentes-Ramos, M., Valor, L.M., del Blanco, B., López-Atalaya, J.P., Barco, A. J Neurosci. 2022 42(42): 7984-8001 https://doi.org/10.1523/JNEUROSCI.0970-22.2022
- Multiomic Analysis of Neurons with Divergent Projection Patterns Identifies Novel Regulators of Axon Pathfinding Fernández-Nogales, M., López-Cascales, M.T., Murcia-Belmonte, V., Escalante, A., Fernández-Albert, J., Muñoz-Viana, R., Barco, A., Herrera, E. Advanced Science 2022 9 (29): art. 2200615 https://doi.org/10.1002/advs.202200615
- Activation-induced chromatin reorganization in neurons depends on HDAC1 activity Grabowska A, Sas-Nowosielska H, Wojtas B, Holm-Kaczmarek D, Januszewicz E, Yushkevich Y, Czaban I, Trzaskoma P, Krawczyk K, Gielniewski B, Martin-Gonzalez A, Filipkowski RK, Olszynski KH, Bernas T, Szczepankiewicz AA, Sliwinska MA, Kanhema T, Bramham CR, Bokota G, Plewczynski D, Wilczynski GM, Magalska A Cell Rep 2022 38(7):110352 https://doi.org/10.1016/j.celrep.2022.110352
- Control of protein synthesis and memory by GluN3A-NMDA receptors through inhibition of GIT1/mTORC1 assembly. Conde-Dusman MJ, Dey PN, Elía-Zudaire Ó, Rabaneda LG, García-Lira C, Grand T, Briz V, Velasco ER, Andero R, Niñerola S, Barco A, Paoletti P, Wesseling JF, Gardoni F, Tavalin SJ, Perez-Otaño I eLife 2021 10:e71575 https://doi.org/10.7554/eLife.71575
- Sublayer- and cell-type-specific neurodegenerative transcriptional trajectories in hippocampal sclerosis Cid E, Marquez-Galera A, Valero M, Gal B, Medeiros DC, Navarron CM, Ballesteros-Esteban L, Reig-Viader R, Morales AV, Fernandez-Lamo I, Gomez-Dominguez D, Sato M, Hayashi Y, Bayés À, Barco A, Lopez-Atalaya JP, de la Prida LM Cell Rep 2021 35(10):109229 https://doi.org/10.1016/j.celrep.2021.109229
- Transcriptome and epigenome analysis of engram cells: Next-generation sequencing technologies in memory research Fuentes-Ramos M, Alaiz-Noya M, Barco A Neurosci Biobehav Rev 2021 127:865 https://doi.org/10.1016/j.neubiorev.2021.06.010
- A Zic2-regulated switch in a noncanonical Wnt/betacatenin pathway is essential for the formation of bilateral circuits Morenilla-Palao C, López-Cascales MT, López-Atalaya JP, Baeza D, Calvo-Díaz L, Barco A, Herrera E Sci Adv 2020 6(46):eaaz8797 https://doi.org/10.1126/sciadv.aaz8797
- KAT3-dependent acetylation of cell type-specific genes maintains neuronal identity in the adult mouse brain Lipinski M, Muñoz-Viana R, Del Blanco B, Marquez-Galera A, Medrano-Relinque J, Caramés JM, Szczepankiewicz AA, Fernandez-Albert J, Navarrón CM, Olivares R, Wilczyński GM, Canals S, Lopez-Atalaya JP, Barco A Nat Commun 2020 11(1):2588 https://doi.org/10.1038/s41467-020-16246-0
- Transcriptome Analysis of iPSC-Derived Neurons from Rubinstein-Taybi Patients Reveals Deficits in Neuronal Differentiation Calzari L, Barcella M, Alari V, Braga D, Muñoz-Viana R, Barlassina C, Finelli P, Gervasini C, Barco A, Russo S, Larizza L Mol Neurobiol 2020 57(9):3685 https://doi.org/10.1007/s12035-020-01983-6
- The Epigenetic Factor CBP Is Required for the Differentiation and Function of Medial Ganglionic Eminence-Derived Interneurons Medrano-Fernandez A, Delgado-Garcia JM, Del Blanco B, Llinares M, Sanchez-Campusano R, Olivares R, Gruart A, Barco A Molecular Neurobiology 2019 56(6):4440 https://doi.org/10.1007/s12035-018-1382-4
- CBP and SRF co-regulate dendritic growth and synaptic maturation Del Blanco B, Guiretti D, Tomasoni R, Lopez-Cascales MT, Muñoz-Viana R, Lipinski M, Scandaglia M, Coca Y, Olivares R, Valor LM, Herrera E, Barco A Cell Death & Differentiation 2019 26(11):2208 https://doi.org/10.1038/s41418-019-0285-x
- CBP/p300 in brain development and plasticity: disentangling the KAT’s cradle Lipinski M, Del Blanco B, Barco A Current Opinion in Neurobiology 2019 59:1 https://doi.org/10.1016/j.conb.2019.01.023
- Cbp-dependent histone acetylation mediates axon regeneration induced by environmental enrichment in rodent spinal cord injury models Hutson TH, Kathe C, Palmisano I, Bartholdi K, Hervera A, De Virgiliis F, McLachlan E, Zhou L, Kong G, Barraud Q, Danzi MC, Medrano-Fernandez A, Lopez-Atalaya JP, Boutillier AL, Sinha SH, Singh AK, Chaturbedy P, Moon LDF, Kundu TK, Bixby JL, Lemmon VP, Barco A, Courtine G, Di Giovanni S Sci Transl Med 2019 11(487):eaaw2064 https://doi.org/10.1126/scitranslmed.aaw2064
- Contribution of spurious transcription to intellectual disability disorders Scandaglia M, Barco A Journal of Medical Genetics 2019 56(8):491 https://doi.org/10.1136/jmedgenet-2018-105668
- Immediate and deferred epigenomic signatures of in vivo neuronal activation in mouse hippocampus Fernandez-Albert J, Lipinski M, Lopez-Cascales MT, Rowley MJ, Martin-Gonzalez AM, Del Blanco B, Corces VG, Barco A Nature Neuroscience 2019 22(10):1718 https://doi.org/10.1038/s41593-019-0476-2
- Early alteration of epigenetic-related transcription in Huntington’s disease mouse models Hervas-Corpion I, Guiretti D, Alcaraz-Iborra M, Olivares R, Campos-Caro A, Barco A, Valor LM Sci Rep 2018 8:9925 https://doi.org/10.1038/s41598-018-28185-4
- Editorial on the Special Issue: Molecules and Cognition Barco A, Knafo S Neuroscience 2018 370:1 https://doi.org/10.1016/j.neuroscience.2017.11.003
- Impact of environmental conditions and chemicals on the neuronal epigenome Del Blanco B, Barco A Curr Opin Chem Biol 2018 45:157 https://doi.org/10.1016/j.cbpa.2018.06.003
- CREB Regulates Distinct Adaptive Transcriptional Programs in Astrocytes and Neurons Pardo L, Valor LM, Eraso-Pichot A, Barco A, Golbano A, Hardingham GE, Masgrau R, Galea E Sci Rep 2017 7:6390 https://doi.org/10.1038/s41598-017-06231-x
- Epigenetic Etiology of Intellectual Disability Iwase S, Berube NG, Zhou Z, Kasri NN, Battaglioli E, Scandaglia M, Barco A J Neurosci 2017 37(45):10773 https://doi.org/10.1523/JNEUROSCI.1840-17.2017
- Loss of Kdm5c Causes Spurious Transcription and Prevents the Fine-Tuning of Activity-Regulated Enhancers in Neurons Scandaglia M, Lopez-Atalaya JP, Medrano-Fernandez A, Lopez-Cascales MT, Del Blanco B, Lipinski M, Benito E, Olivares R, Iwase S, Shi Y, Barco A Cell Rep 2017 21(1):47 https://doi.org/10.1016/j.celrep.2017.09.014
- Revitalizing genetically-modified mouse strains using frozen-thawed sperm after up to 192 h of refrigerated epididymis transportation Moreno-del Val G, Munoz-RobledanoP Lab Anim 2017 51(5):526 https://doi.org/10.1177/0023677217692612
- Targeted activation of CREB in reactive astrocytes is neuroprotective in focal acute cortical injury Pardo L, Schlüter A, Valor LM, Barco A, Giralt M, Golbano A, Hidalgo J, Jia P, Zhao Z, Jove M, Portero-Otin M, Ruiz M, Gimenez-Llort L, Masgrau R, Pujol A, Galea E Glia 2016 64(5):853 https://doi.org/10.1002/glia.22969
- Blocking miRNA Biogenesis in Adult Forebrain Neurons Enhances Seizure Susceptibility, Fear Memory, and Food Intake by Increasing Neuronal Responsiveness Fiorenza A, Lopez-Atalaya JP, Rovira V, Scandaglia M, Geijo-Barrientos E, Barco A Cereb Cortex 2016 26(4):1619 https://doi.org/10.1093/cercor/bhu332
- Nuclear organization and 3D chromatin architecture in cognition and neuropsychiatric disorders Medrano-Fernandez A, Barco A Mol Brain 2016 9:83 https://doi.org/10.1186/s13041-016-0263-x
- Role of Dicer and the miRNA system in neuronal plasticity and brain function Fiorenza A, Barco A Neurobiol Learn Mem 2016 135:3 https://doi.org/10.1016/j.nlm.2016.05.001
- Specific promoter deacetylation of histone H3 is conserved across mouse models of Huntington’s disease in the absence of bulk changes Guiretti D, Sempere A, Lopez-Atalaya JP, Ferrer-Montiel A, Barco A, Valor LM Neurobiol Dis 2016 89:190 https://doi.org/10.1016/j.nbd.2016.02.004
- Fine-tuned SRF activity controls asymmetrical neuronal outgrowth: implications for cortical migration, neural tissue lamination and circuit assembly Scandaglia M, Benito E, Morenilla-Palao C, Fiorenza F, del Blanco B, Coca Y, Herrera E, Barco A Sci Rep 2015 5:17470 https://doi.org/10.1038/srep17470
- The Neuronal Activity-Driven Transcriptome Benito E, Barco A Mol Neurobiol 2015 51(3):1071 https://doi.org/10.1007/s12035-014-8772-z
- Editorial Barco A, Brambilla R, Rosenblum K Neurobiol Learn Mem 2015 124:1 https://doi.org/10.1016/j.nlm.2015.08.001
- cAMP Responsive Element Binding Protein (CREB) and cAMP Co-regulate Activator Protein 1 (AP1)-Dependent Regeneration-Associated Gene Expression and Neurite Growth Ma TC, Barco A, Ratan RR, Willis DE J Biol Chem 2014 289(47):32914 https://doi.org/10.1074/jbc.M114.582460
- Can changes in histone acetylation contribute to memory formation? Lopez-Atalaya J, Barco A Trends Genet 2014 30(12):529 https://doi.org/10.1016/j.tig.2014.09.003
- Epigenetic factors in intellectual disability: the Rubinstein-Taybi syndrome as a paradigm of neurodevelopmental disorder with epigenetic origin Lopez-Atalaya JP, Valor LM, Barco A Prog Molec Biol Transl Sci 2014 128:139 https://doi.org/10.1016/B978-0-12-800977-2.00006-1
- Histone Acetylation and CREB Binding Protein Are Required for Neuronal Resistance against Ischemic Injury Yildririm F, Ji SB, Kronenberg G, Barco A, Olivares R, Benito E, Dirnangl U, Gertz K, Endres M, Harms C, Meisel A PLoS One 2014 9(4):e95465 https://doi.org/10.1371/journal.pone.0095465
- Loss of neuronal 3D chromatin organization causes transcriptional and behavioural deficits related to serotonergic dysfunction Ito S, Magalska A, Alcaraz-Iborra M, Lopez-Atalaya JP, Rovira V,Contreras-Moreira B, Lipinski M, Olivares R, Martinez-Hernandez J,Ruszczycki B, Lujan R,Geijo-Barrientos E,Wilczynski GM, Barco A Nat Commun 2014 5:4450 https://doi.org/10.1038/ncomms5450
- Neuroepigenetic disorders: Progress, promises and challenges. Barco A Neuropharmacology 2014 80:1 https://doi.org/10.1016/j.neuropharm.2014.02.013
- Histone H3 lysine methylation in cognition and intellectual disability disorders Parkel S, Lopez-Atalaya JP, Barco A Learn Mem 2013 20(10):570 https://doi.org/10.1101/lm.029363.112
- Genomic landscape of transcriptional and epigenetic dysregulation in early-onset polyglutamine disease Valor LM, Guiretti D, Lopez-Atalaya JP, Barco A J Neurosci 2013 33(25):10471 https://doi.org/10.1523/JNEUROSCI.0670-13.2013
- Lysine acetyltransferases CBP and p300 as therapeutic targets in cognitive and neurodegenerative disorders Valor LM, Viosca J, Lopez-Atalaya JP, Barco A Curr Pharm Design 2013 19(28):5051
- Genomic targets, and histone acetylation and gene expression profiling of neural HDAC inhibiton Lopez-Atalaya JP, Ito S, Valor LM, Benito E, Barco A Nucleic Acids Res 2013 41(17):8072 https://doi.org/10.1093/nar/gkt590
- Histone acetylation deficits in lymphoblastoid cell lines from patients with Rubinstein-Taybi syndrome Lopez-Atalaya JP, Gervasini C, Mottadelli F, Spena S, Piccione M, Scarano G, Selicorni A, Barco A, Larizza L J Med Genet 2012 49(1):66 https://doi.org/10.1136/jmedgenet-2011-100354
- Hippocampal gene profiling: Toward a systems biology of the hippocampus Valor LM, Barco A Hippocampus 2012 22(5):929 https://doi.org/10.1002/hipo.20888
- Enhanced cAMP Response Element-Binding Protein Activity Increases Neuronal Excitability, Hippocampal Long-Term Potentiation, and Classical Eyeblink Conditioning in Alert Behaving Mice Gruart A, Benito E, Delgado-Garcia JM, Barco A J Neurosci 2012 32(48):17431 https://doi.org/10.1523/JNEUROSCI.4339-12.2012
- Genetic Approaches to Investigate the Role of CREB in Neuronal Plasticity and Memory Barco A, Marie H Mol Neurobiol 2011 44(3):330 https://doi.org/10.1007/s12035-011-8209-x
- CBP is required for environmental enrichment-induced neurogenesis and cognitive enhancement Lopez-Atalaya JP, Ciccarelli A, Viosca J, Valor LM, Jimenez-Minchan M, Canals S, Giustetto M, Barco A EMBO J 2011 30(20):4287 https://doi.org/10.1038/emboj.2011.299
- cAMP Response Element-Binding Protein Is a Primary Hub of Activity-Driven Neuronal Gene Expression Benito E, Valor JM, Jimenez-Minchan M, Huber W, Barco A J Neurosci 2011 31(50):18237 https://doi.org/10.1523/JNEUROSCI.4554-11.2011
- Ablation of CBP in Forebrain Principal Neurons Causes Modest Memory and Transcriptional Defects and a Dramatic Reduction of Histone Acetylation But Does Not Affect Cell Viability Valor LM, Pulopulos MM, Jimenez-Minchan M, Olivares R, Lutz B, Barco A J Neurosci 2011 31(5):1652 https://doi.org/10.1523/JNEUROSCI.4737-10.2011
- Ultrastructural and transcriptional profiling of neuropathological misregulation of CREB function Valor LM, Jancic D, Lujan R, Barco A Cell Death Differ 2010 17(10):1636 https://doi.org/10.1038/cdd.2010.40
- Syndromic features and mild cognitive impairment in mice with genetic reduction on p300 activity: Differential contribution of p300 and CBP to Rubinstein-Taybi syndrome etiology Viosca J, Lopez-Atalaya JP, Olivares R, Eckner R, Barco A Neurobiol Dis 2010 37(1):186 https://doi.org/10.1016/j.nbd.2009.10.001
- CREB’s control of intrinsic and synaptic plasticity: implications for CREB-dependent memory models Benito E, Barco A Trends Neurosci 2010 33(5):230 https://doi.org/10.1016/j.tins.2010.02.001
2009
- Sanchis-Segura C*, Lopez-Atalaya JP* and Barco A (2009). Selective boosting of transcriptional and behavioral responses to drugs of abuse by histone deacetylase inhibition. Neuropsychopharmacology 34(13): 2642-54. * Equal contributors. (Featured article )
- Sanchis-Segura C, Jancic D, Jimenez-Minchan M and Barco A (2009). Inhibition of cAMP responsive element binding protein in striatal neurons enhances approach and avoidance responses towards morphine- and morphine withdrawal-related cues. Front Behav Neurosci 3:30 Open access frontiers
- Viosca J *, Lopez de Armentia M *, Jancic D and Barco A (2009). Enhanced CREB-dependent gene expression increases the excitability of neurons in the basal amygdala and primes the consolidation of contextual and cued fear memory. Learn Mem 16(3): 193-7.* Equal contributors
- Viosca J, Malleret G, Bourtchouladze R, Benito E, Vronskava S, Kandel ER and Barco A (2009). Chronic enhancement of CREB activity in the hippocampus interferes with the retrieval of spatial information. Learn Mem 16(3): 198-209 (issue cover).
- Jancic D*, Lopez de Armentia M*, Valor LM, Olivares R and Barco A (2009). Inhibition of cAMP-response element binding protein reduces neuronal excitability and plasticity, and triggers neurodegeneration. Cereb Cortex 19(11): 2535-47. * Equal contributors.
- Barco A (2009). Circuitos moleculares implicados en la formación de recuerdos. Chapter in La demencia: epidemia que crece (Ed. Fundación Neurociencias y Envejecimiento).
- Viosca J, Schuhmacher A, Guerra and Barco A (2009). Germ line expression of H-RasG12V causes neurological deficits associated to Costello syndrome. Genes Brain Behav 8(1): 60-71.
2008
- Barco A, Lopez de Armentia M and Alarcon JM (2008). Synapse-specific stabilization of plasticity processes: The synaptic tagging hypothesis revisited ten years later. Neurosci Biobehav Rev 32(4):831-51.
- Hawkins RD, Bailey H, Barco A and Kandel ER (2008). Molecular studies of learning and memory in Aplysia and the hippocampus: A comparative analysis of implicit and explicit memory storage. Chapter in “Learning and memory: A comprehensible reference” (Elsevier Books).
2007
- Lopez de Armentia M,Jancic D, Olivares R, Alarcon ER, Kandel ER and Barco A (2007).CREB-mediated gene expression increases the intrinsic excitability of CA1 pyramidal neurons. J Neurosci 27(50): 13909-13918.
- Barco A, Jancic D and Kandel ER (2007). CREB-dependent transcription and synaptic plasticity. Chapter in “Regulation of transcription by neuronal activity: To the nucleus and back” (Springer Science publishers).
- Barco A and Lopez de Armentia M (2007). El cerebro plástico. Chapter in Unidad didáctica “Viaje al Universo Neuronal”(FECYT).
- Barco A (2007). The Rubinstein-Taybi syndrome: Modeling mental impairment in the mouse. Genes, Brain & Behavior 6(S1):32-39.
2006
- Gasic GP, Barco A, Avila J and Lerma J (2006). A meeting to remember: Meeting on memory and related disorders. EMBO Reports 7(8):768-73.
- Barco A, Bailey CH and Kandel ER (2006). Common molecular mechanisms in explicit and implicit memory. J Neurochem 97(6):1520-33.
- Alarcon JM, Barco A and Kandel ER (2006). Capture of L-LTP within and across the apical and basilar dendritic compartments of CA1 pyramidal neurons: synaptic tagging is restricted to specific dendritic compartments. J Neurosci 26(1):256-264
2005
- Barco A and Kandel ER (2005). Role of CREB and CBP in brain function. Chapter in Transcription factors in the nervous system: Development, brain function and disease (Wiley-VCH publishers).
- Levine AA, Guan Z, Barco A, Xu S, Kandel ER and Schwartz JH (2005). CREB binding protein controls response to cocaine by acetylating histones at the fosB promoter in the mouse striatum. Proc Natl Acad Sci U S A. 102(52):19186-91.
- Barco A, Patterson S, Alarcon JM, Gromova P, Mata-Roig M, Morozov A and Kandel ER (2005). Gene expression profiling of facilitated L-LTP in VP16-CREB mice reveals that BDNF is critical for both the maintenance of LTP and for synaptic capture. Neuron, 48(1): 123-137.
2004
- Calandria C, Irurzun A, Barco A and Carrasco L (2004).Individual expression of poliovirus 2APRO induces caspase-3 activation and PARP cleavage in HeLa cells. Virus Research 104(1): 39-49.
- Alarcon JM, Malleret G, Touzani K, Vronskaya S, Ishii S, Kandel ER and Barco A (2004). Chromatin acetylation, memory, and LTP are impaired in CBP+/- mice: a model for the cognitive deficit in Rubinstein-Taybi syndrome and its amelioration. Neuron 42(6):947-959 (issue cover) (Featured article )
- Pham TA, Graham SJ, Suzuki S, Barco A, Kandel ER, Gordon B and Lickey ME (2004). A semi-persistent adult ocular dominance plasticity in visual cortex is stabilized by activated CREB. Learning and memory 11(6): 738-747.
- Gao Y, Deng K, Hou J, Bryson JB, Barco A, Nikulina E, Spencer T, Mellado W, Kandel ER and Filbin MT (2004). Activated CREB is sufficient to overcome inhibitors in myelin and promote spinal axon regeneration in vivo. Neuron 44(4):609-621.
- Lang C, Barco A, Zablow L, Kandel R, Siegelbaum SA, Zakharenko S (2004). Transient expansion of synaptically connected dendritic spines upon induction of hippocampal long-term potentiation. Proc Natl Acad Sci U S A. 101(47):16665-70.
2003
- Barco A, Pittenger C and Kandel ER (2003).CREB, memory enhancement and the treatment of memory disorders: promises, pitfalls and prospects. Expert Opinion on Therapeutic Targets 7(1):101-14.
2002
- Barco A, Alarcon JM and Kandel ER (2002) Expression of constitutively active CREB protein facilitates the late phase of long-term potentiation by enhancing synaptic capture. Cell 108(5):689-703.
- Agirre A, Barco A, Carrasco L and Nieva JL (2002) Viroporin-mediated membrane permeabilization: pore formation by non-structural poliovirus 2B protein. J Biol Chem 277(43):40434-40441.
- Carrasco L, Guinea R, Irurzun A and Barco A. (2002) Effects of viral replication on cellular membrane metabolism and function. Chapter in «MolecularBiology of Picornavirus» edited by B.L. Semler and E. Wimmer (ASM Press)
2000
- Barco A, Feduchi E and Carrasco L. (2000) Poliovirus Protease 3C(pro) Kills Cells by Apoptosis. Virology 266:352-360.
- Barco A, Feduchi E and Carrasco L. (2000) A stable HeLa cell line that inducibly expresses poliovirus 2Apro: effects on cellular and viral gene expression. J Virol 74: 2383-2392.
1999
- Ventoso I, Barco A and Carrasco L. (1999) Genetic selection of poliovirus 2Apro-binding peptides. J Virol 73:814-818.
1998
- Barco A. and Carrasco L. (1998) Identification of regions involved in cytotoxicity in poliovirus 2BC protein. J. Virol. 72: 3560-3570.
- Barco A. and Carrasco L. (1998) Co-expression of human eIF-4G and poliovirus 2Apro in Saccharomyces cereviseae Effects on gene expression. J Gen Virol 79:2651-60.
- Ventoso I, Barco A and Carrasco L. (1998) Mutational analysis of poliovirus 2Apro distinct inhibitory functions of 2Apro on translation and transcription. J Biol Chem 273:27960-27967.
1997
- Barco A, Ventoso I and Carrasco L. (1997). The yeast Saccharomyces cerevisiae as a genetic system for obtaining variants of poliovirus protease 2A. J Biol Chem 272: 12683-12691.
1996
- Aldabe R, Barco A and Carrasco L. (1996).Membrane permeabilization by poliovirus protein 2B and 2BC. J Biol Chem 271: 23134-23137.
1995
- Barco A and Carrasco L. (1995). Cloning and inducible synthesis of poliovirus non-structural proteins in Saccharomyces cerevisiae. Gene 156: 19-25.
- Barco A and Carrasco L. (1995). A human virus protein, poliovirus protein 2BC, induces membrane proliferation and blocks the exocytic pathway in the yeast Saccharomyces cereviseae. The EMBO J 14: 3349-3364.
- Barco A and Carrasco L. (1995). Poliovirus 2Apro expression inhibits growth of yeast cells. FEBS Letters 371: 4-8.
Alumni, visitors and former members:
- Samanta Ortuño graduada en Biotecnología en la Universidad Miguel Hernández de Elche (UMH), con los Másteres en Biotecnología y Bioingeniería (UMH) y en Bioinformática (Universidad de Murcia). Realizó el trabajo de fin de Grado y Máster en biología molecular en el laboratorio del Dr. Antonio Vera (UMH). Continuó su experiencia trabajando en Metabarcoding y Bioinformática en la empresa Taxon Estudios Ambientales S.L. y en el laboratorio del Dr. José Antonio García Charton (Universidad de Murcia).
- Ana Martín estudió Biología en la Universidad de Málaga. Gracias al apoyo de una beca internacional de doctorado Marie Curie fellowship, Ana está desarrollando su proyecto de doctorado en el marco de una colaboracion entre el grupo del Prof. Wilczynski en el Nencki Institute of Experimental Biology (Varsovia) y nuestro laboratorio.
- Mayte López Cascales es licenciada en Bioquímica por la Universidad de Murcia, con un Máster de Neurociencias por la Universidad Pablo de Olavide de Sevilla, y Máster de Bioinformática en la Universidad de Murcia. En 2014 recibió el premio al mejor trabajo fin de master de bioinformatica por la universidad de Murcia. Actualmente está realizando su tesis doctoral en el grupo de la Dra. Herrera y trabaja en varios proyectos colaborativos entre los dos laboratorios.
- Jordi Fernández studied Biology at the Universitat de Girona (UdG) and got his Interuniversity Master degree in Neuroscience coordinated by the Universitat de Barcelona (UB). He was first trained in neurosciences and in molecular and cell biology techniques in the laboratories of Dr. Gemma Huguet (School of Science, UdG), Dr. Carles Sindreu (School of Medicine, UB) in which he conducted his master thesis, and Dr. Manuel Irimia (Centre de Regulació Genòmica, CRG). He joined our group in February 2015 thanks to a fellowship awarded by the Severo Ochoa program (MINECO). He defended his Ph.D. thesis in October 2019 and obtained the maximal qualification. Thesis: Immediate and deferred epigenomic signatures of in vivo neuronal activation in mouse hippocampus. Publications with us: Fernandez-Albert et al., Nat Neurosci 22, 1718-30; Lipinski et al., Nat Comm 11(1):2588. Other positions: Lab. Dr. Mark Schnitzer. University of Stanford, San Francisco, San Francisco, USA.
- Michal Lipinski estudió Biotecnología en la Universidad de Varsovia de Ciencias de la Vida (Polonia) y ha realizado estancias en laboratorios del Instituto Nencki de Biología Experimental (Varsovia) y del Departamento de Anatomía de la Universidad de Zurich. En Junio del 2012 se incorporó a nuestro grupo para realizar su tesis doctoral gracias a una beca financiada por el programa de Becas Santiago Grisolia de la Generalitat Valenciana. He defended his Ph.D. thesis in December 2019 and obtained the maximal qualification. Thesis: Role of CBP and p300 in the establishment and maintenance of transcriptional programs in adult excitatory neurons. Publications with us: Ito et al., Nat Comm 5:4450; Galvao-Ferreira et al., eNeuro 4(2); Scandaglia et al. Cell Reports 21(1):47-59; del Blanco et al., Cell Death Differ 26(11):2208-2222; Lipinski et al., Curr Opin Neurobiol 59:1-8;Fernandez-Albert et al., Nat Neurosci 22, 1718-30; Lipinski et al., Nat Comm 11(1):2588. Other positions: Lab. Dr. Paola Arlotta & Aviv Regev. Harvard Department of Stem Cell and Regenerative Biology, Cambridge & BROAD Institute. MA, USA.
- Marilyn Scandaglia studied Molecular Biotechnology at the Università Alma Mater Studiorum di Bologna in Italy. In 2011, as a pregraduate student, she moved to the Instituto de Neurociencias de Alicante and joined our lab to carry out her Master and PhD theses. She defended her Ph.D. thesis in January 2018 and obtained the maximal qualification. She also received the special distinction (Premio Extraordinario). Thesis: Role of lysine demethylase 5C in neurodevelopment and intellectual disability. Publications with us: Fiorenza et al., Cereb Cortex 26(4):1619-33; Scandaglia et al., Sci Reports5:17470; Iwase et al., J Neurosci 37(45):10773-10782; Scandaglia et al. Cell Reports 21(1):47-59; del Blanco et al., Cell Death Differ 26(11):2208-2222; Scandaglia and Barco. J Med Gen 56(8):491-498. Other positions: Lab. Dr. Berta Lopez Sanchez-Laorden. Instituto de Neurociencias (UMH-CSIC), Alicante, Spain.
- Anna Fiorenza studied Biology at the Università degli studi del Sannio in Italy. In 2008, she moved to the Instituto de Neurociencias de Alicante, as part of the «Erasmus Placement» subprogram in the U.E. Lifelong Learning Programme, where she worked in the lab of Dr. Lerma in the physiology of kainate receptors. In 2010, she was awarded a fellowship from the INA-Consolider Program and decided to join our group to carry out her thesis. She defended her Ph.D. thesis in September 2015 and obtained the maximal qualification. Thesis: Regulation of neuronal plasticity and responsiveness by the microRNA system. Publications with us: Fiorenza et al., Cereb Cortex 26(4):1619-33; Scandaglia et al., Sci Reports5:17470; Fiorenza & Barco, Neurobiol Learn Mem May 135:3-12. Other positions: Lab. Dr. Dorit Ron. University of California, San Francisco, San Francisco, USA; Institute for Neurodegenerative Diseases, University of California, San Francisco, San Francisco, USA.
- Deisy Guiretti studied Biology at the National University of Misiones, Argentina. She later investigated in the field of viral variability and phylogenia at the National Institute of Cancer in Rio de Janeiro and in the University of Edinburgh. She joined our lab in 2009 to carry out her thesis working on the etiology of Huntington’s disease. She defended her Ph.D. thesis in September 2015 and obtained the maximal qualification. Thesis: Lysine deacetylation and transcriptional dysregulation in neurological disorders: Huntington’s disease and the Rubinstein-Taiby syndrome. Publications with us: Valor et al., J Neurosci 33(25):10471-82; Guiretti D and Valor LM, Neuropharmacology 80:103-114; Guiretti et al., Neurobiol Dis 89:190-201; Hervas-Corpion et al., Sci Rep 8(1):9925; del Blanco et al., Cell Death Differ 26(11):2208-2222.
- Jose Viosca got his Master degree in Biochemistry in the Universidad de Valencia. He worked with us from October 2005 to May 2010 investigating the role of CREB and neuronal chromatin acetylation in learning and memory. He defended his Ph.D. thesis in May 2012 and obtained the maximal qualification. He also received the special distinction (Premio Extraordinario). He later moved to the laboratory of Cornelius Gross in EMBL-Monterotondo (Italy) for his first postdoctoral stay. Thesis: Common molecules in memory formation and congenital intellectual disability: a role for the Ras-ERK-CREB pathway in cognition. Publications with us: Viosca et al., J Neurosci 27(47): 12761; Viosca et al. Learn Mem16(3): 193; Viosca et al. Learn Mem 16(3): 198; Viosca et al. Genes Brain Behav 8(1):60; Viosca et al. Neurobiol Dis 37(1):186; Lopez-Atalaya et al. EMBO J 30(20):4287; Valor et al. Curr Pharm Des 19(28):5051-64. Other positions: Lab. Dr. Cornelius Gross. EMBL Monterotondo. Monterotondo, Italy.
- Eva Benito-Garagorri studied Biochemistry at the University of the Basque Country. She carried out her PhD in our laboratory working on the transcriptional response to neuronal activity and defended her Ph.D. thesis in June 2011, obtaining the maximal qualification. She is currently a postdoc at the laboratory of Andre Fischer in ENI-Gottingen (Germany). Thesis: A comparative transcriptomics approach for unveiling gene expression networks of activity-driven neuronal stimulation and plasticity. Publications with us: Viosca et al., J Neurosci 27(47): 12761; Viosca et al., Learn Mem 16(3): 198; Benito and Barco, Trends Neurosci 33(5): 230; Benito et al., J Neurosci 31(50): 18237; Gruart et al., J Neurosci 32(48): 17431; Lopez-Atalaya et al., Nucleic Acids Res. 41(17):8072-84; Yildirim F et al. Plos ONE 9(4); Benito and Barco. Mol Neurobiol. 289(47):32914-25; Scandaglia et al., Sci Reports 5:17470; Scandaglia et al. Cell Reports 21(1):47-59. Other positions: Lab. Dr. Andrè Fischer. DZNE, Gottingen, Germany. Scientific Coordinator for SourceData at EMBO, Heidelberg, Germany.
- Dragana Jancic studied Medicine and got her MD in the University of Belgrade (Serbia). She became a member of the Marie Curie Excellence Team in November 2004 and worked with us until July 2008. She defended her Ph.D. thesis in December 2008 and obtained the maximal qualification. She is currently a resident in the Psychiatry and Psychotherapy Department at the University Hospitals of Geneva (Switzerland). Thesis: Role of CREB-dependent transcription in the control of hippocampal neurons survival and plasticity. Publications with us: Barco et al. chapter in “Regulation of transcription by neuronal activity: To the nucleus and back” (Springer Sci Pub); Viosca et al. J Neurosci27(47): 12761; Lopez de Armentia et al. J Neurosci27(50): 13909; Viosca et al. Learn Mem 16(3): 193; Jancic et al. Cereb Cortex 19(11): 2535; Sanchis-Segura et al. Front Behav Neurosci3:30; Valor et al. Cell Death Differ 17(19): 1636. Other positions: Resident in Psychiatry and Psychotherapy, University Hospitals of Geneva. Geneva, Switzerland.
Other former lab members:
– Juan Medrano: Graduate student. He carried out his postgraduate studies from January 2017 to July 2020.
– Dr. Rafael Muñoz Viana: Posdoctoral fellow. He was part of the Team from June 2017 to December 2019.
– Marian Llinares: Technician. She was part of the Team from January 2018 to December 2019.
– Emanuele Zaccaria: Graduate student. He was part of the Team from June 2018 to June 2019.
– Alejandro Medrano: Graduate student. He carried out his Master thesis with us in 2015 and his postgraduate studies from January 2016 to July 2018.
– Nuria Cascales Pico: Technician. She was part of the Team from Julio 2016 to November 2017.
– Dr. Jose P. López-Atalaya: Senior postdoctoral fellow and Senior Researcher. He was part of the Team from 2006 to September 2015. He is now Principal Investigator at our Institute leading the «Cellular Plasticity and Neuropathology» group.
– Dr. Romana Tomasoni: Posdoctoral fellow and Visiting Researcher. She joined our lab from 2015 to 2016.
– Dr. Luis M. Valor: Posdoctoral fellow, Senior Researcher and finally co-PI. He was part of the Team from August 2007 to November 2015. He is now Principal Investigator at Hospital Universitario Puerta del Mar in Cadiz, Spain.
– Manuel Alcaraz Iborra: Graduate student. He was part of the Team from June 2012 to January 2014. He then went to the Psychology Department at University of Almeria (Spain) to conduct there his Ph.D. thesis.
– Victor Rovira: After finishing his thesis in the team of Emilio Geijo-Barrientes, he joined the Team from June to December 2013 before moving to , USA for a postdoc in the laboratory of Manuel Castro-Alamancos at Drexel University, Philadelphia, USA.
– Dr. Sven Parkel: Postdoctoral fellow. He was part of the Team from January 2012 to June 2013.
– Dr. Satomi Ito: Postdoctoral fellow. She was part of the Team from April 2009 to March 2013. She later worked at the Institute for Integrated Cell-material Sciences (iCeMS) in Kyoto University and the Okinawa Institute of Science anf Technology (OIST, Ichiro Maruyama’s lab), both in Japan.
– Pierrick Jego:Graduate student. He was part of the Team from October 2010 to September 2011.
– Francisca Almagro: Technician. She was part of the Team from November 2010 to July 2011.
– María Jiménez-Minchan: Technician. She was part of the Team from August 2008 to June 2011.
– Matías M Pulopulos: Graduate student. He was part of the Team from October 2009 to August 2010.
– Dr. Mikel Lopez de Armentia: Senior Researcher. He was part of the Team from November 2004 to August 2009.
– Miguel A. Andrés: Graduate student. He was part of the Team from July 2007 to December 2008.
– Marusa Arencibia: Secretary. She was our administrative officer from 2005 to 2008.
– Valentina Moscato: Graduate student. She worked as a member of the MC Excellence Team form September 2007 to April 2008.
– Petra Gromova: Graduate student. She was in the lab from November 2004 to July 2007.
– Ana Calvo: Graduate student. She was part of the Team from October 2005 to March 2006.
Visitors:
– María Consuelo López Gómez (undergraduate student, Master en Neurociencias UMH). Octubre 2019 – Enero 2021
– Pablo Castellano (undergraduate student, Master en Neurociencias UPO). May-October 2019
– Miguel Fuentes Ramos (undergraduate student, Universidad de Alicante, Alicante). Octubre 2017 – Septiembre 2018
– Marta Alaiz Noya (undergraduate student, Master en Neurociencias UMH). Octubre 2017 – Septiembre 2018
– Paula Mut (undergraduate student, Master en Neurociencias UMH). Octubre 2016 – Septiembre 2017
– Juan Marín (undergraduate student, Master en en Bioinformática y BiologÃa Computacional de la Escuela Nacional de salud- ISCIII). May-October 2019
– Juan Medrano (undergraduate student, Universidad Jaume I, Castellón). July-September 2015
– Agnieszka Czechwska (undergraduate student, Warsaw University, Warsaw, Poland). July-August 2015
– Silvia Colmenero (undergraduate student, Universidad de Alicante, Alicante). may-September 2015
– Alejandro Medrano (undergraduate student, Universidad de Alicante, Alicante). June-October 2014
– Juan Manuel Agulló (undergraduate student, Universidad Autónoma de Barcelona, Barcelona). July-August 2014
– Adriana Magalska (postdoctoral fellow, Dr. Wilczynski’s lab, Nencki Institute, Warsaw, Poland). September to December 2013
– Marilyn Scandaglia (undergraduate student, University of Bologna, Bologna, Italy). April-October 2011
– Jorge Montesinos (undergraduate student, Universidad de Valencia, Valencia). Augus-September 2010
– Raffaela Tulino (undergraduate student, University of Sannio, Sannio, Italy). April-September 2010
– Alessandro Ciccarelli(PhD student, Dr. Giustetto’s lab, University of Torino, Torino, Italy). July 2009 to June 2010
– Paola Tognino (PhD student, Dr. Pizzorusso’s lab, CNR Pisa, Italy). February 2010
– Gianmarco di Mauro (undergraduate student, University of Bologna, Bologna, Italy). May-December 2009
– Vincent Prinz (postdoctoral fellow, Dr. Dirnagl’s lab, Charite Campus Mitte, Berlin, Germany). September 2009
– Jan-Albert Manenschijn (rotation student, Alicante). April 2009
– Eduardo Leiva (rotation PhD student, Alicante). February-March 2009
– Elena Carnero (undergraduate student, Universidad de Granada, Granada). Summer 2008
– Ferah Yildirim (PhD student, Dr. Dirnagl’s lab, Charite Campus Mitte, Berlin, Germany). July 2008
– Oskar Ortiz (PhD student, Dr. Moratalla’s lab, Instituto Cajal, Madrid). April 2008
– Carles Sanchis-Segura (Ramón y Cajal researcher, Universitat Jaume I, Castellon). October-November 2007
– Pankaj Sah (University Professor, University of Queensland, St. Lucia, Australia). September-October 2007
– Cristina Vicente (undergraduate student, Universidad Pablo de Olavide, Sevilla). Summer 2007
– Ignasi Sahun (PhD student, Dr. Dierssens lab,CRG, Barcelona). November 2005

 English
English